

| En el campo de la ciencia aprendemos cuán grande es la extrañeza del mundo | Robert Oppenhimer |
Un plásmido es una pequeña molécula de ADN circular que a menudo se encuentra en las bacterias y otras células.
Los plásmidos se utilizan como vectores de clonación por su capacidad de reproducirse de manera independiente al ADN cromosómico y porque es relativamente fácil
manipularlos e insertar nuevas secuencias genéticas.
Los plásmidos juegan un papel determinante en la diseminación de la resistencia a antibióticos, son moléculas de ADN que replican de forma independiente al
cromosoma bacteriano y tienen la capacidad de movilizar genes horizontalmente.
Un enzima de restricción es aquel que puede reconocer una secuencia característica de nucleótidos dentro de una molécula de ADN y cortar el ADN en ese punto
concreto llamado diana de restricción o en un sitio no muy lejano a este.
Los enzimas de restricción se suelen usar para insertar genes y otros fragmentos de ADN en plásmidos durante la clonación de ADN. Son producidos por las
bacterias como un mecanismo de defensa contra las infecciones víricas, concretamente contra virus bacteriófagos cuya reproducción depende de la maquinaria
genética de la bacteria.
- Tubos eppendorf.
- Baño termoregulado.
- Micropipeta.
- Enzimas de restricción:
- Eco RI
- Hind III
- Bam HI
- Tampón adecuado para cada enzima.
- Plásmidos:
- pTurboFP635
- pHAF1
- Colorantes:
- SimplySafeTM: colorante que emite fluorescencia al ser iluminado con luz LED azul.
- Azul de bromofenol.
- Gel de agarosa al 0,7%
- Matraz Erlenmeyer.
- Cubeta de electroforesis.
- Tampón de electroforesis TBE 1X (Tris, Borato, EDTA).
- LUZ LED Azul.
1°. Se utilizará agarosa al 0,7% en tampón 1xTBE. Pesamos 0,21 gramos de agarosa en un matraz Erlenmeyer, añadimos 30mL de tampón 1xTBE y fundimos
en un horno microondas. Preparamos el recipiente para el gel sellando por los lados una bandeja de electroforesis con cinta adhesiva.
2°. Cuando el matraz se encuentre templado, añadimos 2µL de colorante fluorescente SimplySafeTM. Vertemos la mezcla en la bandeja y colocamos el
peine cerca de uno de los bordes de la misma. Dejamos que la agarosa solidifique sin mover la bandeja. El colorante se intercalará con las bases
del DNA y emitirá fluorescencia en el rango visible del espectro electromagnético, tras ser iluminado con luz LED azul, permitiendo visualizar los
fragmentos de DNA.
3°. Una vez solidificado el gel, retiramos el peine y observamos los pocillos formados. Retiramos las cintas adhesivas. Lo colocamos en la cubeta
de electroforesis sumergiéndolo en tampón 1xTBE. Si no vamos a utilizar el gel hasta el día siguiente, lo envolvemos en un plástico y lo dejamos en
la nevera hasta entonces.
 |
| Imagen con parte del material que utilizaremos en el trabajo. |
1°. Tanto las enzimas como los plásmidos deben de ser conservados en hielo ya que de no estar en un frío constante se degradarían. La digestión se realizará con:
- 1 µL de plásmidos.
- 1 µL de enzimas.
- 2 µL de buffer.
- 16 µL de H2O
2°. Todo esto lo llevaremos a un baño María durante aproximadamente 1h 30 minutos.
 | 
|
| Baño maría durante 1h 30min. | |
Una vez transcurrido el tiempo de digestión de la muestra de DNA plasmídico, añadimos, antes de cargarla en el gel, 2µL de tampón de carga de electroforesis.
Este tampón contiene:
- Glicerol: Un agente densificante que sirve para que las muestras sedimenten en el fondo de los pocillos.
- Azul de bromofenol: Colorante de pequeño tamaño molecular que sirve para controlar la migración del frente de electroforesis.
 |
| Añadimos 2µL de tampón de carga de electroforesis. |
1°. Colocamos cuidadosamente cada muestra en un pocillo distinto del gel mediante una micropipeta automática. Reservamos un pocillo para colocar una muestra de Marker III (20µL) que contiene los fragmentos de DNA de tamaño conocido (patrones). 2°. Finalmente, conectamos los electrodos de la cubeta de electroforesis a la fuente de alimentación, aplicando un voltaje de 48V durante una hora.
 | 
|
| Preparamos la muestra para la electroforesis. | |
Una vez terminada la electroforesis, colocamos el gel sobre un vidrio con fondo negro y se ilumina con luz LED azul para observar los fragmentos y fotografiarlos. Para una correcta fotografía, es recomendable utilizar un filtro naranja.
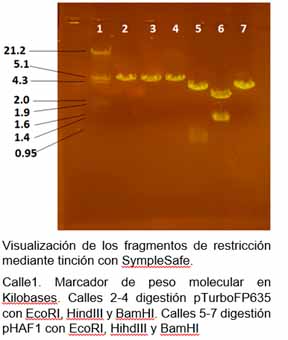
En esta digestión de plásmidos, tras haber llevado acabo la electroforesis y la tinción con un colorante fluorescente, se pueden observar los fragmentos por
los cortes que el enzima de restricción ha hecho sobre los plásmidos.
El primer plásmido (pTurboFP635) se cortó con 3 enzimas de restricción (Eco RI, Hind III, Bam HI) originando una banda en cada
caso, respectivamente. Con el patrón observado, este plásmido presenta un único punto de corte o diana, originando un solo fragmento de alrededor de 5,1Kb
(kilobases).
En las siguientes 3 calles, el plásmido pHAF1 es cortado por los mismos enzimas que el anterior. En este caso, los resultados observados son más variados.
En la calle 5, con el enzima Eco RI, se ven dos fragmentos, el mayor de aproximadamente 3,5 Kb y el menor alrededor de 0.95 Kb. En la quinta calle se
originan dos fragmentos por la acción del enzima Hind III, el mayor de aproximadamente 2 Kb y el menor de aproximadamente 1,8 Kb. Estos resultados
indican que el plásmido pHAF1 tiene dos dianas para los enzimas Eco RI y Hind III. Por último, en la sexta calle se visualiza un único fragmento en la digestión
por el enzima Bam HI de algo más de 3.5 Kb. Este resultado, indica que sólo presenta una sola diana de restricción.
 |